大上研究室のYoutube Channelです。研究紹介や国際会議の発表動画などが掲載されています。

研究事例
- タンパク質間相互作用予測システムMEGADOCKの開発
- 網羅的なPPI予測データベースMEGADOCK-Webの開発
- 膜タンパク質のタンパク質間相互作用予測技術の開発
- 超高速タンパク質間ドッキング計算による構造生物学応用
- AlphaFold2などのタンパク質立体構造予測の利活用
- 低分子化合物の薬効・活性・ADMET予測
- 「薬らしさ」を表現する統計モデルの開発
- 「PPI阻害薬らしさ」を表現する統計モデルの開発
- ランク学習によるLBDDスクリーニング法の開発
- 深層学習による薬剤標的相互作用予測
- 薬剤標的相互作用予測のApplicability Domainに関する研究
- 創薬至適ペプチドのスクリーニングのための技術開発
- AlphaFold2によるタンパク質-ペプチド複合体解析と配列設計
- AlphaFold2によるタンパク質-環状ペプチド複合体予測
- AlphaFold2の併用による構造ベースバーチャルスクリーニング
- TSUBAME 4.0やABCIによるGPUスーパーコンピューティング技術の開発
- 「富岳」による超並列計算応用
- 結合自由エネルギー計算による高活性分子の推定
- 結合自由エネルギー計算の並列計算技術に関する研究
- 能動学習を利用した結合自由エネルギー計算の効率化
- 創薬に適した核酸配列設計に関する研究
- 天然物や天然物類縁体の生物活性予測
- 抗体設計支援技術の開発
上記は一例です。大上研ではバイオインフォマティクスや創薬分野に係る多くのテーマを扱っています。
タンパク質間相互作用予測システムMEGADOCKの開発、応用
タンパク質の立体構造情報を利用して、タンパク質間相互作用 (protein-protein interaction, PPI) を予測するソフトウェアを開発しています。
世の中のソフトウェアと比較してGPU実装やマルチノード並列化が進んでおり、高速に大量のタンパク質間相互作用を予測することができます。(産総研ABCIスパコン512ノードで、1日あたり約7000万PPIを予測できます。)

MEGADOCK version 4は非営利利用を目的としてCC BY-NCライセンスで公開されています。商用利用の際はご相談ください。
Githubリポジトリはこちら https://github.com/akiyamalab/MEGADOCK
Google Colaboratoryで試してみたい方はこちら https://github.com/ohuelab/MEGADOCK-on-Colab
MEGADOCKは、NVIDIA GPU Application Catalogにも掲載されています。 
MEGADOCKによるPPI研究の応用事例にはたとえば以下のものがあります。
Kami D, Kitani T, Nakamura A, Wakui N, Mizutani R, Ohue M, Kametani F, Akimitsu N, Gojo S.
The DEAD-box RNA-binding protein DDX6 regulates parental RNA decay for cellular reprogramming to pluripotency.
PLoS ONE, 13(10): e0203708, 2018. doi:10.1371/journal.pone.0203708
![]() Journal website | PubMed
Journal website | PubMed
Hayashi T, Matsuzaki Y, Yanagisawa K, Ohue M, Akiyama Y.
MEGADOCK-Web: an integrated database of high-throughput structure-based protein-protein interaction predictions.
BMC Bioinformatics, 19(Suppl 4): 62, 2018. doi:10.1186/s12859-018-2073-x
![]() Journal website | PubMed | Slide
Journal website | PubMed | Slide
大上雅史, 林孝紀, 秋山泰.
タンパク質間相互作用と複合体構造の予測結果を検索できるウェブサイト「MEGADOCK-Web」.
実験医学, 2019年6月号, 37(9): 1469-1474, 羊土社, 2019.
Book website
ペプチド創薬支援
低分子医薬品・抗体医薬品につぐ第3の医薬品として注目を集めるペプチド医薬品に対して、分子の性質を解明するための研究や、創薬を支援する計算技術を開発しています。
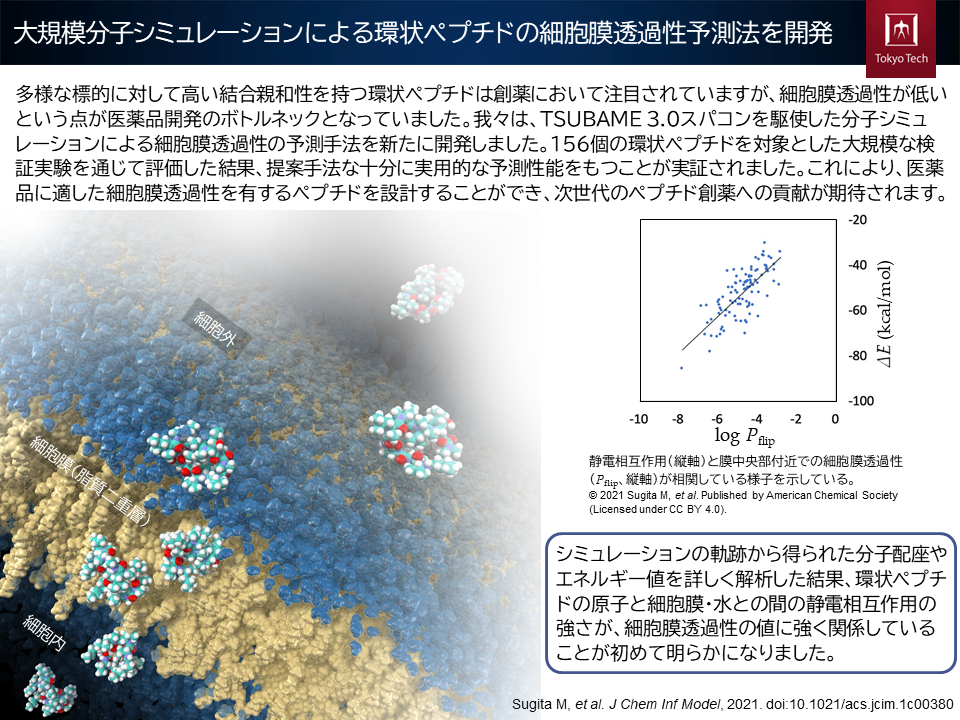
ペプチド創薬のための分子設計手法として、環状ペプチドの細胞膜透過性を予測する計算手法を開発しました。拡張サンプリング法を用いた分子動力学シミュレーションによる予測手法を考案し、約150個の環状ペプチドによる大規模な検証によってその精度が実証されました。環状ペプチドの膜透過性は医薬品設計におけるボトルネックとなっており、本研究がペプチド創薬に大きく貢献するものと期待しています。(文部科学省 地域イノベーション・エコシステム形成プログラムの成果)
 Sugita M, Fujie T, Yanagisawa K, Ohue M, Akiyama Y.
Sugita M, Fujie T, Yanagisawa K, Ohue M, Akiyama Y.
Lipid composition is critical for accurate membrane permeability prediction of cyclic peptides by molecular dynamics simulations.
Journal of Chemical Information and Modeling, 62(18): 4549-4560, 2022. doi: 10.1021/acs.jcim.2c00931
![]() Journal website | PubMed
Journal website | PubMed
 Sugita M, Sugiyama S, Fujie T, Yoshikawa Y, Yanagisawa K, Ohue M, Akiyama Y.
Sugita M, Sugiyama S, Fujie T, Yoshikawa Y, Yanagisawa K, Ohue M, Akiyama Y.
Large-scale membrane permeability prediction of cyclic peptides crossing a lipid bilayer based on enhanced sampling molecular dynamics simulations.
Journal of Chemical Information and Modeling, 61(7): 3681-3695, 2021. doi: 10.1021/acs.jcim.1c00380
![]() Journal website | PubMed
Journal website | PubMed
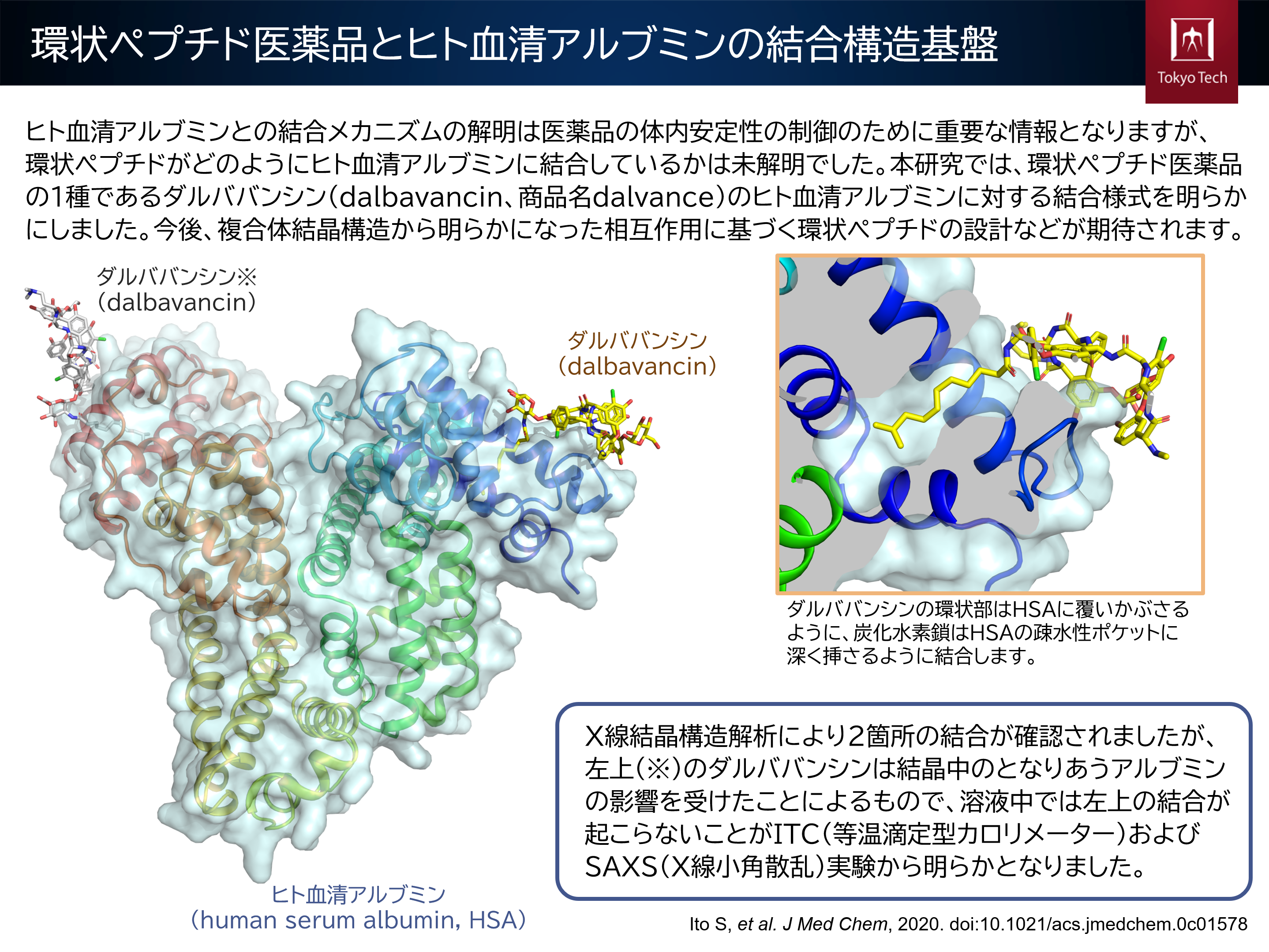
環状ペプチド医薬品の体内安定性の向上のため、これまでに報告されていなかったヒト血清アルブミンとの複合体構造を新規に報告しました。これにより、環状ペプチドがどのようにヒト血清アルブミンに結合して体内安定性を保持するかが明らかとなりました(長岡工業高等専門学校、株式会社リガク、東京大学との共同研究成果)。
Ito S, Senoo A, Nagatoishi S, Ohue M, Yamamoto M, Tsumoto K, Wakui N.
Structural basis for binding mechanism of human serum albumin complexed with cyclic peptide dalbavancin.
Journal of Medicinal Chemistry, 63(22): 14045–14053, 2020. doi:10.1021/acs.jmedchem.0c01578
![]() Journal website | bioRxiv | PubMed
Journal website | bioRxiv | PubMed
競争的研究資金
- 文部科学省 科学研究費助成事業(科学研究費補助金)学術変革領域研究(A) 計画研究(代表)
「bRO5化合物の潜在空間構築と応用のための情報科学」
2023年4月~2028年3月、60,060,000円(総額) - 文部科学省 科学研究費助成事業(科学研究費補助金)学術変革領域研究(A) 計画研究(総括班)(分担)
「天然物が織り成す化合物潜在空間が拓く生物活性分子デザイン」
2023年4月~2028年3月(研究代表者:菊地和也) - 日本学術振興会 科学研究費助成事業(科学研究費補助金)基盤研究(B)(代表)
「環状ペプチドの複合体構造データベース構築と分子設計技術の開発」
2023年4月~2026年3月、18,850,000円(総額) - 日本学術振興会 科学研究費助成事業(科学研究費補助金)基盤研究(B)(分担)
「スリット膜裏打ち蛋白MAGI-2の機能解析と新規CKD治療薬の開発」
2023年4月~2026年3月(研究代表者:淺沼克彦) - 国立研究開発法人 科学技術振興機構(JST) 創発的研究支援事業(代表)
「マルチモダリティ創薬を拓くインフォマティクス基盤」
2022年4月~2025年3月、31,460,000円(総額) - 国立研究開発法人日本医療研究開発機構(AMED) 生命科学・創薬研究支援基盤事業(BINDS)(分担)
「スーパーコンピュータ資源及び大規模シミュレーションとAIに基づく創薬・生命科学の支援」
2022年4月~2028年3月(研究代表者:関嶋政和) - 日本学術振興会 科学研究費助成事業(学術研究助成基金助成金)基盤研究(C)(分担)
「野放図なAIを創薬ドメイン知識で使いこなす化合物最適化支援ツール基盤の構築」
2022年4月~2025年3月(研究代表者:山本一樹) - 国立研究開発法人 科学技術振興機構(JST) 戦略的創造研究推進事業 ACT-X(代表)※終了しました
「タンパク質を制御するペプチドのデザインAI」
2020年12月~2023年3月&加速フェーズ2023年4月~2024年3月、16,458,000円(総額) - 日本学術振興会 科学研究費助成事業(科学研究費補助金)基盤研究(B)(代表)※終了しました
「タンパク質間相互作用を標的とする薬剤分子設計技術の開発」
2020年4月~2023年3月、17,680,000円(総額) - 文部科学省 地域イノベーション・エコシステム形成プログラム(分担)※終了しました
「IT創薬技術と化学合成技術の融合による革新的な中分子創薬フローの事業化」
2017年9月~2022年3月(事業プロデューサー:舛屋 圭一) - 国立研究開発法人日本医療研究開発機構(AMED) 創薬等先端技術支援基盤プラットフォーム(BINDS)(分担)※終了しました
「大規模分子シミュレーションによるインシリコスクリーニング支援と構造インフォマティクス技術の高度化」
2017年4月~2022年3月(研究代表者:関嶋政和) - 公益財団法人 みずほ学術振興財団 第63回工学研究助成(代表)※終了しました
「生体分子構造の高速な類似度計算手法の開発」
2020年4月~2022年3月、2,000,000円(総額) - 公益財団法人 上原記念生命科学財団 研究奨励金(代表)※終了しました
「創薬至適化合物を選出する深層ランク学習」
2020年1月~2021年4月、2,000,000円(総額)
企業との共同研究
(公表されているもの or 許可を得て掲載するもの)
- モジュラス株式会社 東京工業大学との共同研究契約締結のお知らせ(プレスリリース)
https://prtimes.jp/main/html/rd/p/000000006.000035986.html - ぺプチドリーム株式会社 特殊ペプチド創薬向けインシリコ技術の共同研究契約を締結(プレスリリース)
https://www.titech.ac.jp/news/2016/035981.html
